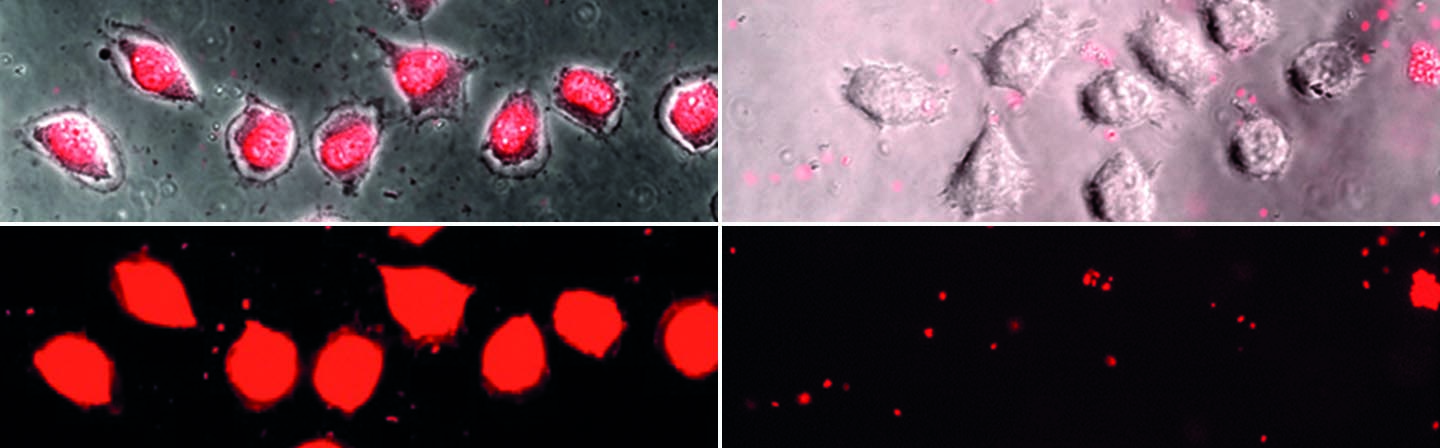
Inaktivierungsstrategien zur Keimlastreduzierung (AMPs)
Mit Hilfe von antimikrobiellen Peptiden (AMPs) können Oberflächen mit einer bioziden Wirkung ausgestattet werden. Dafür werden nicht-toxische AMPs, synthetischen oder natürlichen Ursprungs, auf den jeweiligen Materialien immobilisiert. Durch die Anwendung der AMPs in Medizinprodukten, wie z.B. in Wundauflagen, kann der Einsatz von Antibiotika eingeschränkt werden. Die Keimlast wird somit reduziert ohne drohende Resistenzen zu fördern.
Leistungsangebot:
- Kopplung von Peptiden an gewünschte Oberfläche mittels affiner oder kovalenter Bindung
- Kopplung über »Hilfsmatrices« auf schwierigen Substraten
 Fraunhofer-Institut für Zelltherapie und Immunologie, Institutsteil Bioanalytik und Bioprozesse IZI-BB
Fraunhofer-Institut für Zelltherapie und Immunologie, Institutsteil Bioanalytik und Bioprozesse IZI-BB