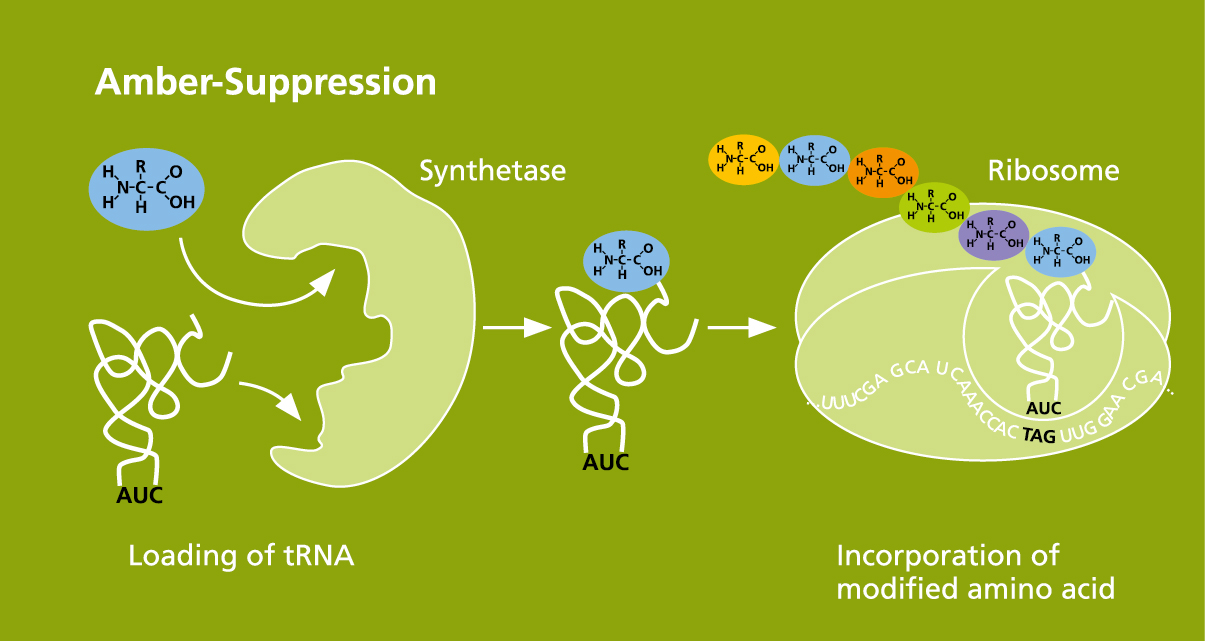
Von Natur aus existieren 20 Standardaminosäuren aus denen, abgesehen von wenigen Ausnahmen, jedes Protein besteht. Aufgrund dieser limitierenden Anzahl an Aminosäuren ergeben sich somit Einschränkungen in den biochemischen und biophysikalischen Eigenschaften gewünschter funktioneller Proteine.

Die Arbeitsgruppe »Zellfreie Proteinsynthese« bietet neben der reinen Synthese auch eine verlässliche Methode um chemisch modifizierte Aminosäuren mit verschiedensten reaktiven Gruppen an ortsspezifischen Positionen in die zellfrei synthetisierten Proteinen einzuführen. Als Grundlage dafür wird die Methodik der amber-Suppression genutzt. Des Weiteren ermöglichen wir über Click-Chemie die Kopplung von Fluoreszenzfarbstoffen, Zuckerstrukturen, PEGylierungen und Biotinylierungen an die reaktiven Gruppen der eingeführten Aminosäuren. Besonders für die Strukturanalyse und Funktionsbestimmung von Membranproteinen, aber auch für das Screening von neuartigen Liganden und Therapeutika kann unsere Methode genutzt werden. Außerdem können Proteine auf diese Weise selektiv verändert werden, sodass sich neue Funktionen für therapeutische Zwecke ergeben können.
Die veränderten Proteine können für Strukturanalysen und Funktionsbestimmungen oder ein Screening neuartiger Liganden eingesetzt werden oder durch neue Funktionalitäten auch für therapeutische Zwecke interessant werden.
 Fraunhofer-Institut für Zelltherapie und Immunologie, Institutsteil Bioanalytik und Bioprozesse IZI-BB
Fraunhofer-Institut für Zelltherapie und Immunologie, Institutsteil Bioanalytik und Bioprozesse IZI-BB