Die Produktion von rekombinanten Proteinen stellt eines der Kernthemen der heutigen Biotechnologie und der pharmazeutischen Industrie dar. Jedes Protein benötigt individuell adaptierte Produktionsprozesse, um neben höchster Qualität auch eine maximale Ausbeute gewährleisten zu können. Unser Institut verfügt über langjährige Expertise in der Produktion rekombinanter Proteine und der zugehörigen Prozessoptimierung. Neben der konventionellen, zellbasierten Produktion liegt unsere Spezialisierung vor allem in der zellfreien Proteinsynthese. Diese Methode ermöglicht nicht nur eine schnellere Produktionszeit und macht den Syntheseprozess ökonomisch, der Prozess wird auch flexibler und besser steuerbar. Außerdem ermöglicht diese Technologie die Produktion von sonst schwer herstellbaren Proteinen, wie Membranproteinen oder zytotoxisch wirkenden Proteinen.
Proteinsynthese

Möglichkeiten für zellbasierte Produktionsprozesse am Fraunhofer IZI-BB. Diverse Möglichkeiten der Kultivierung von eukaryotischen Zellen von Schüttelkolben bis Benchtop (1 l und 5 l) (Bühnenbild - Pilotscale 30l) Fermentationsprozessen. Die Auswahl der Kultivierungsart wird an die jeweilige Anwendung und Entwicklung angepasst.
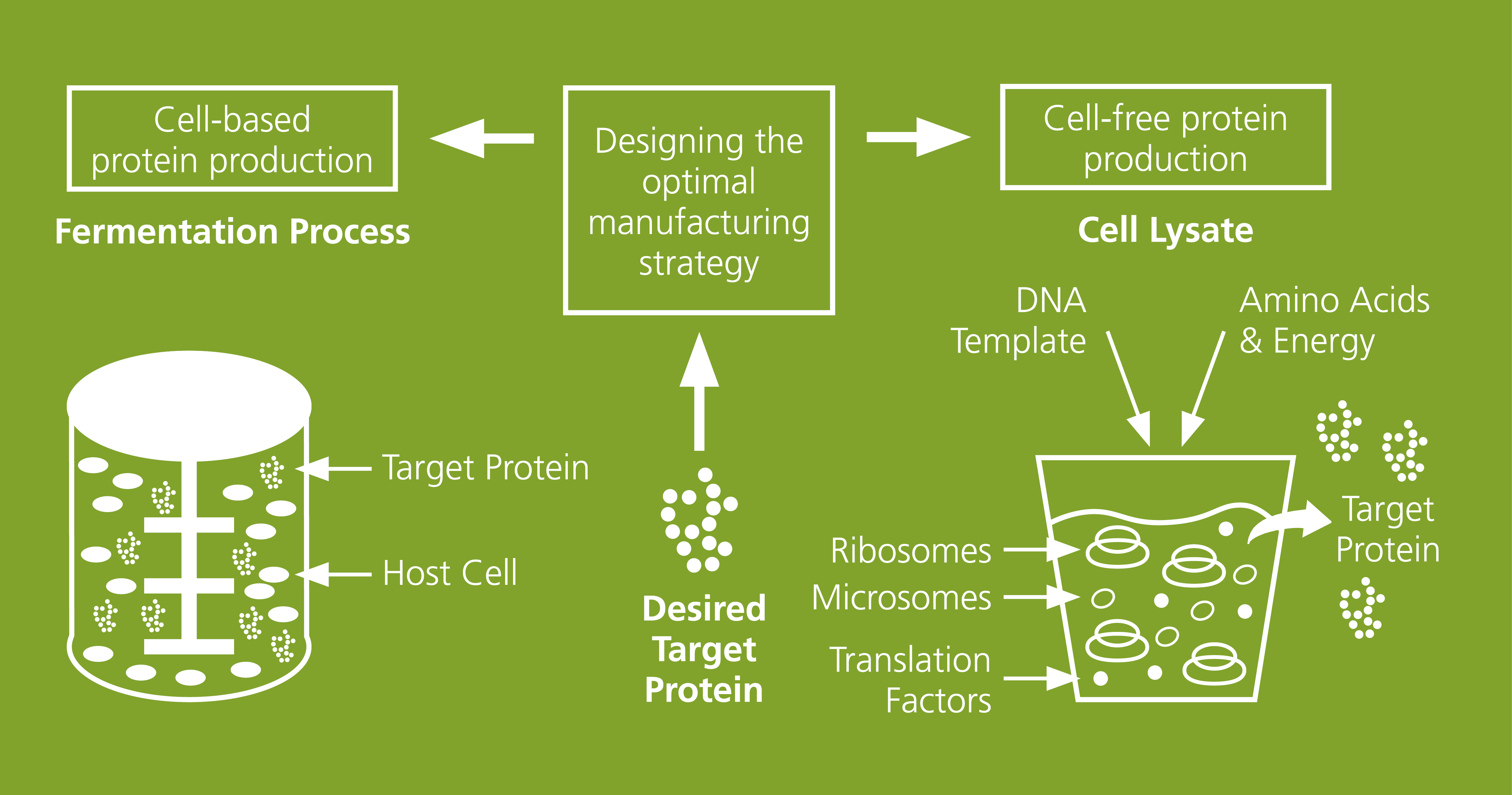
Entwicklung und Evaluierung von Proteinproduktionsprozessen angepasst an die individuellen Bedingungen eines erwünschten Zielproteins. Am Fraunhofer IZI-BB sind neben konventionellen, zellbasierten Proteinproduktionssystemen (linker Teil der Abbildung) spezialisierte zellfreie Proteinsyntheseplattformen (rechter Teil der Abbildung) vorhanden, die eine schnelle und parallelisierte Synthese von Proteinen ermöglichen. Die Auswahl der Methode erfolgt abhängig von der zukünftigen Anwendung und den Charakteristiken der Proteine.
Leistungsangebote:
- »in vivo« versus »in vitro« (zellbasiert versus zellfrei): Vergleiche von Gensequenzen, Promotersequenzen, Signalpeptiden und Reinheitsmarkern für eine Verbesserung und Beschleunigung von industriellen Produktionspipelines
Zellbasierte Synthese
- Nutzung von eukaryotischen Zelllinien (CHO,humane Zelllinien, Sf21) zur rekombinanten Proteinproduktion
- Evaluation verwendeter Transfektionsmethoden zum Erhalt von hohen Expressionsraten von Zielproteine
- Entwicklung stabiler Zelllinien für gewünschte Prozesse mittels verschiedener Klonselektions- und Genintegrationsmethoden (z.B. CRISPR Cas)
- Hochskalierung von Fermentationsprozessen für die rekombinante Proteinproduktion bis zum Pilotmaßstab
Zellfreie Synthese
- Zellfreie Synthese von Membranproteinen und toxischen Proteinen
- Nanodisks zur Einbettung von Membranproteinen
- Schnelle, funktionelle Analyse von Ionenkanälen, Transporterproteinen und porenbildenen, toxischen Proteine direkt nach der Synthese durch elektrophysiologische Methoden in planaren Doppellipidschichten
- Maßgeschneiderte Zellfrei-Systeme für kundenspezifische Anwendungen
- Prokaryotische zellfreie Systeme basierend auf E.coli - Lysaten
- Eukaryotische zellfreie Systeme basierend auf CHO, Sf21 und K562 Zelllysaten
- Hochskalierung der Zellfreisysteme für die Produktion und Aufreinigung von medizinisch relevanten Proteinen
- Entwicklung neuer HTS-Methoden und HTS-kompatibler Systeme für Screening-Anwendungen
- Kundenspezifische Anpassung von Zelllinien für maßgeschneiderte Zellfrei-Systeme
Anwendungen
Reiternavigation
Methoden
- Herstellung, Optimierung und Klonierung von »ready-to-express« DNA-Templaten für die zellfreie Proteinsynthese
- RNA-Synthese (Transkription, Analyse und Aufreinigung von mRNA)
- Zellfreie Synthese und Charakterisierung rekombinanter Antikörperformate
- Synthese rekombinanter Antikörperformate auf Basis linearer oder zirkulärer DNA-Template in eukaryotischen In-vitro-Transkriptions-Translationssystemen
- Parallele Herstellung der Antikörperformate in verschiedenen Reaktionsführungen, z.B. im Batch- und Dialyse-Modus bzw. gekoppelt (Transkription und Translation in einem Reaktionssystem) und entkoppelt (Transkription und Translation in separaten Reaktionen)
- Bestimmung der Syntheseausbeute mittels (14C)-Protein-Labeling und TCA-Präzipitation
- Charakterisierung der Proteinexpression durch Gelelektrophorese, Autoradiographie und quantitatives Imaging im Phosphorimager
- Protein-Analyse mittels Fluoreszenz-Mikroskopie und Western Blotting
- Gerichtete Proteinevolution durch Mutagenese und Aktivitäts-Screening
- Kotranslationale Markierung von Antikörperfragmenten, z.B. mit Fluoreszenzfarbstoffen
- Funktionsuntersuchungen an zellfrei hergestellten Antikörperfragmenten z.B. durch ELISA
- Optimierung von In-vitro-Translationssystemen zur Synthese Disulfid-verbrückter Proteine
Geräte und Anlagen
Zellkulturlabore der Sicherheitsklasse S1
- 5 L Fermenter (Sartorius Biostat B DCU-II Advanced Additive Flow System; 2x 5 l Kessel, aufrüstbar mit bis zu 6 Gefäßen zwischen 1 und 10 l)
- 30 L Fermenter (Sartorius Biostat D DCU)
- konfokales Laserscanning-Mikroskop (Zeiss CLSM 510)
- automatisierte Zellassay- und Screening-Einheit (PerkinElmer; CellLuxCellularFluorescence Workstation)
Isotopenlabor
- Proteinmarkierung (Umgang mit 14C, 32P, 35S)
- Absauganlage für die Abtrennung von 14C-markierten Proteinpräzipitaten (TCA-Präzipitation)
- Szintillationszähler (Beckmann LS 6500 Multi Purpose Scintillation Counter)
- Geltrocknungsanlage für Autoradiogramme (Unigeldryer 3545)
- Typhoon Trio+ variable-mode-Imager (Radioaktivität, Fluoreszenz und Chemilumineszenz mit erweitertem 10 µm Pixel-Scan)
Labore der Sicherheitsklasse S1 für molekularbiologische Arbeiten
- Multimode Reader Berthold LB 941 vi-S TriStar (Flash-, Glow- und Farb-Lumineszenz, Absorption, Fluoreszenz, FRET, BRET)
- Sirius single tube Luminometer (Titertek Berthold)
- Spektralphotometer für UV/Vis (Nanodrop ND-2000c)
- Bioreaktoren für die zellfreie Proteinsynthese im Batchverfahren und im Dialysemaßstab
Massenspektrometrie in Laboren der Sicherheitsklasse S1
- Massenspektrometer Q-TOF MaxIs Impact (Bruker Daltonics) mit austauschbaren Ionenquellen (offline nanoESI source, conventional ESI source, captive spray für die Nano-LC Kopplung)
- Ultra-sensitive Ionenfalle AmaZon Speed ETD (Bruker Daltonics) mit austauschbaren Ionenquellen (conventional ESI source, captive spray für die Nano-LC Kopplung)
- UHPLC Chromatographieanlagen, Ultimate 3000 nanoRSLC system (Dionex)
Analytik von Membranproteinen in Laboren der Sicherheitsklasse S1
- Patch-Clamp System »Port-A-Patch« und Orbit 16 der Firma Nanion
- Partikelanalyse mittels »Zetasizer Nano ZS« der Firma Malvern
Publikationen und Patente
- Ullrich, J.; Göhmann, P. J.; Zemella, A.; Kubick, S. (2022) Oligomerization of the heteromeric γ-aminobutyric acid receptor GABAB in a eukaryotic cell-free system. Scientific reports. 10.1038/s41598-022-24885-0.
- Schloßhauer, J. L.; Cavak, N.; Zemella, A.; Thoring, L.; Kubick, S. (2022) Cell Engineering and Cultivation of Chinese Hamster Ovary Cells for the Development of Orthogonal Eukaryotic Cell-free Translation Systems. Frontiers in Molecular Biosciences. 10.3389/fmolb.2022.832379.
- Ramm, F.; Jack, L.; Kaser, D.; Schloßhauer, J. L.; Zemella, A.; Kubick, S. (2022) Cell-Free Systems Enable the Production of AB5 Toxins for Diagnostic Applications. Toxins. 10.3390/toxins14040233.
- Ramm, F.; Dondapati, S. K.; Trinh, H. A.; Wenzel, D.; Walter, R. M.; Zemella, A.; Kubick, S. (2022) The Potential of Eukaryotic Cell-Free Systems as a Rapid Response to Novel Zoonotic Pathogens: Analysis of SARS-CoV-2 Viral Proteins. Frontiers in Bioengineering and Biotechnology. 10.3389/fbioe.2022.896751.
- Knauer, J. F.; Liers, C.; Hahn, S.; Wuestenhagen, D. A.; Zemella, A.; Kellner, H.; Haueis, L.; Hofrichter, M.; Kubick, S. (2022) Cell-free production of the bifunctional glycoside hydrolase GH78 from Xylaria polymorpha. Enzyme and Microbial Technology. 10.1016/j.enzmictec.2022.110110.
- Walter, R. M.; Zemella, A.; Schramm, M.; Kiebist, J.; Kubick, S. (2022) Vesicle-based cell-free synthesis of short and long unspecific peroxygenases. Frontiers in Bioengineering and Biotechnology. 10.3389/fbioe.2022.964396.
- Haueis, L.; Stech, M.; Kubick, S. (2022) A Cell-free Expression Pipeline for the Generation and Functional Characterization of Nanobodies. Frontiers in Bioengineering and Biotechnology. 10.3389/fbioe.2022.896763.
- Krebs, S. K.; Stech, M.; Jorde, F.; Rakotoarinoro, N.; Ramm, F.; Marinoff, S.; Bahrke, S.; Danielczyk, A.; Wüstenhagen, D. A.; Kubick, S. (2022) Synthesis of an Anti-CD7 Recombinant Immunotoxin Based on PE24 in CHO and E. coli Cell-Free Systems. International Journal of Molecular Sciences. 10.3390/ijms232213697.
- Krebs, S. K.; Rakotoarinoro, N.; Stech, M.; Zemella, A.; Kubick, S. (2022) A CHO-Based Cell-Free Dual Fluorescence Reporter System for the Straightforward Assessment of Amber Suppression and scFv Functionality. Frontiers in Bioengineering and Biotechnology. https://doi.org/10.3389/fbioe.2022.873906.
- Ramm, F.; Stech, M.; Zemella, A.; Frentzel, H.; Kubick, S. (2021) The Pore-Forming Hemolysin BL Enterotoxin from Bacillus cereus: Subunit Interactions in Cell-Free Systems. Toxins. 10.3390/toxins13110807.
- Stech, M.; Rakotoarinoro, N.; Teichmann, T.; Zemella, A.; Thoring, L.; Kubick, S. (2021) Synthesis of Fluorescently Labeled Antibodies Using Non-Canonical Amino Acids in Eukaryotic Cell-Free Systems. Methods in molecular biology (Clifton, N.J.). 10.1007/978-1-0716-1406-8_9.
- Dhandapani, P.; Dondapati, S. K.; Zemella, A.; Bräuer, D.; Wüstenhagen, D. A.; Mergler, S.; Kubick, S. (2021) Targeted esterase-induced dye (TED) loading supports direct calcium imaging in eukaryotic cell-free systems. RSC advances. 10.1039/d0ra08397f.
- WÜSTENHAGEN, D.A., LUKAS, P., Müller, C., Aubele, S.A., Hildebrandt, J-P., KUBICK, S.: „Cell-free Synthesis of the hirudin variant 1 of the blood-sucking leech Hirudo medicinalis” Sci Rep 10, 19818 (2020). https://doi.org/10.1038/s41598-020-76715-w
- Dondapati, S.K., STECH, M., M., ZEMELLA, A., KUBICK, S.: Cell‑Free Protein Synthesis: A Promising Option for Future Drug Development. BioDrugs (2020), https://doi.org/10.1007/s40259-020-00417-y
- RAMM, F., DONDAPATI, S.K., THORING L., ZEMELLA A., WÜSTENHAGEN, D.A., FRENTZEL, H., STECH M., KUBICK, S.: “Mammalian cell-free protein expression promotes the functional characterization of the tripartite non-hemolytic enterotoxin from Bacillus cereus”. Sci Rep. 10, 2887 (2020).
- ZAITSEVA, E., DONDAPATI, S., SCHLOßHAUER, J., ZEMELLA, A., DHANDAPANI, P., KUBICK, S., BAAKEN, G.: „Functional Characerization of Ion Channels Expressed in Eukaryotic Cell-free Systems using Lipid Bilayer Arrays”. Biophysical Journal (2020), Volume 118, Issue 3, Supplement 1, 586A.Dondapati, S.K., STECH, M., M., ZEMELLA, A., KUBICK, S.: Cell‑Free Protein Synthesis: A Promising Option for Future Drug Development. BioDrugs (2020), https://doi.org/10.1007/s40259-020-00417-y
- RAMM, F., DONDAPATI, S.K., THORING L., ZEMELLA A., WÜSTENHAGEN, D.A., FRENTZEL, H., STECH M., KUBICK, S.: “Mammalian cell-free protein expression promotes the functional characterization of the tripartite non-hemolytic enterotoxin from Bacillus cereus”. Sci Rep. (2020) 10:2887.
- Jérôme V, Thoring L, Salzig D, Kubick S, Freitag R. Comparison of cell-based versus cell-free mammalian systems for the production of a recombinant human bone morphogenic growth factor. Engineering in Life Sciences 2017 Aug 7. doi:10.1002/elsc.201700005
- Zemella A, Grossmann S, Sachse R, Sonnabend A, Schaefer M, Kubick S. Qualifying a eukaryotic cell-free system for fluorescence based GPCR analyses. Sci Rep. 2017 Jun 16;7(1):3740. doi:10.1038/s41598-017-03955-8. Artikel
- Georgi V, Georgi L, Blechert M, Bergmeister M, Zwanzig M, Wüstenhagen DA, Bier FF, Junga E, Kubick S. On-chip automation of cell-free protein synthesis: new opportunities due to a novel reaction mode. Lab Chip. 2016 Jan 5;16(2):269-81. DOI dx.doi.org/10.1039/c5lc00700c. Artikel
- Quast RB, Ballion B, Stech M, Sonnabend A, Varga BR, Wüstenhagen DA, Kele P, Schiller SM, Kubick S. Cell-free synthesis of functional human epidermal growth factor receptor: Investigation of ligand-independent dimerization in Sf21 microsomal membranes using non-canonical amino acids. Sci Rep. 2016 Sep 27;6:34048. DOI dx.doi.org/10.1038/srep34048 Artikel
- Quast RB, Sonnabend A, Stech M, Wüstenhagen DA, Kubick S. High-yield cell-free synthesis of human EGFR by IRES-mediated protein translation in a continuous exchange cell-free reaction format. Sci Rep. 2016 Jul 26;6:30399. DOI dx.doi.org/10.1038/srep30399 Artikel
- Thoring L, Wüstenhagen DA, Borowiak M, Stech M, Sonnabend A, Kubick S. Cell-Free Systems Based on CHO Cell Lysates: Optimization Strategies, Synthesis of »Difficult-to-Express« Proteins and Future Perspectives. PLoS One. 2016 Sep 29;11(9):e0163670. DOI dx.doi.org/10.1371/journal.pone.0163670 Artikel
- Zemella A, Thoring L, Hoffmeister C, Kubick S. Cell-Free Protein Synthesis: Pros and Cons of Prokaryotic and Eukaryotic Systems. Chembiochem. 2015 Oct 19. DOI dx.doi.org/10.1002/cbic.201500340. [Epub ahead of print] Artikel
- Quast RB, Mrusek D, Hoffmeister C, Sonnabend A, Kubick S. Cotranslational incorporation of non-standard amino acids using cell-free protein synthesis. FEBS Lett. 2015 Jul 8;589(15):1703-12. DOI dx.doi.org/10.1016/j.febslet.2015.04.041. Epub 2015 May 1. Artikel
- Bechlars S, Jäckel C, Diescher S, Wüstenhagen DA, Kubick S, Dieckmann R, Strauch E. Characterization of trh2 Harbouring Vibrio parahaemolyticus Strains Isolated in Germany. PLOS ONE | DOI dx.doi.org/10.1371/journal.pone.0118559. Artikel
- Quast RB, Kortt O, Henkel J, Srujan KD, Wüstenhagen DA, Stech M, Kubick S. Automated production of functional membrane proteins usingeukaryotic cell-free translation systems. Journal of Biotechnology 203 (2015) 45–53. Artikel
- Brödel AK, Wüstenhagen DA, Kubick S. Cell-Free Protein Synthesis Systems Derived from Cultured Mammalian Cells. Methods Mol Biol. 2015;1261:129-40. doi: 10.1007/978-1-4939-2230-7_7.
- Stech M, Kubick K. Cell-Free Synthesis Meets Antibody Production: A Review. Antibodies 2015, 4, 12-33; DOI dx.doi.org/10.3390/antib4010012.
- Dondapati SK, Kreir M, Quast RB, Wüstenhagen DA, Brüggemann A, Fertig N, Kubick S. Membrane assembly of the functional KcsA potassium channel in a vesicle-based eukaryotic cell-free translation system. Biosens Bioelectron. 2014 Sep 15;59:174-83.
- Sachse R, Dondapati SK, Fenz SF, Schmidt T, Kubick S. Membrane protein synthesis in cell-free systems: from bio-mimetic systems to bio-membranes. FEBS Letter. 2014 Aug 25;588(17):2774-81. DOI dx.doi.org/10.1016/j.febslet.2014.06.007
- Stech M, Hust M, Schulze C, Dübel S, Kubick S. Cell-free eukaryotic systems for the production, engineering, and modification of scFv antibody fragments. Engineering in Life Sciences. 2014;14(4):387–398. DOI dx.doi.org/10.1002/elsc.201400036.
- Stech M, Quast RB, Sachse R, Schulze C, Wüstenhagen DA, Kubick S. A continuous-exchange cell-free protein synthesis system based on extracts from cultured insect cells. PLoS One 9 (2014) e96635.
- Scheller FW, Yarman A, Bachmann T, Hirsch T, Kubick S, Renneberg R, Schumacher S, Wollenberger U, Teller C, Bier FF. Future of biosensors: a personal view. Adv Biochem Eng Biotechnol. 2014;140:1-28.
- Stech M, Brödel AK, Quast RB, Sachse R, Kubick S. Cell-free systems: functional modules for synthetic and chemical biology. Advances in Biochemical Engineering/Biotechnology, Springer Berlin Heidelberg (2013) 67-102.
- Quast RB, Claussnitzer I, Merk H, Kubick S, Gerrits M. Synthesis and site-directed fluorescence labeling of azido proteins using eukaryotic cell-free orthogonal translation systems. Anal Biochem. 2014 Apr 15;451:4-9.
- Fenz SF, Sachse R, Schmidt T, Kubick S. Cell-free synthesis of membrane proteins: tailored cell models out of microsomes. Biochimica et Biophysica Acta. 2014 May;1838(5):1382-8. DOI dx.doi.org/10.1016/j.bbamem.2013.12.009.
- Sachse R, Wüstenhagen D, Samal?kova M, Gerrits M, Bier FF, Kubick S. Synthesis of membrane proteins in eukaryotic cell-free systems. Eng. Life Sci. 2013, 13, No. 1, 39–48.
- Bechlars S, Wüstenhagen DA, Drägert K, Dieckmann R, Strauch E, Kubick S. Cell-free synthesis of functional thermostable direct hemolysins of Vibrio parahaemolyticus. Toxicon. 2013 Dec 15;76:132-42.
- Brödel AK, Sonnabend A, Kubick S. Cell-free protein expression based on extracts from CHO cells. Biotechnol Bioeng. 2014 Jan;111(1):25-36.
- Brödel AK, Sonnabend A, Roberts L, Stech M, Wüstenhagen DA, Kubick S. IRES-Mediated translation of membrane proteins and glycoproteins in eukaryotic cell-free systems. PLoS One 8 (2013) e82234.
- Brödel AK, Raymond JA, Duman JG, Bier FF, Kubick S. Functional evaluation of candidate ice structuring proteins using cell-free expression systems. J Biotechnol. 2013 Feb 10;163(3):301-10.
- Stech M, Merk H, Schenk JA, Stöcklein W, Wüstenhagen DA, Micheel B, Duschl C, Bier FF, Kubick S. Production of functional antibody fragments in a vesicle-based eukaryotic cell-free translation system. J Biotechnol. 164 (2012) 220-231.
- Zampatis DE, Rutz C, Furkert J, Schmidt A, Wüstenhagen D, Kubick S, Tsopanoglou NE, Schülein R. The protease-activated receptor 1 possesses a functional and cleavable signal peptide which is necessary for receptor expression. FEBS Lett. 2012 Jul 30;586(16):2351-9.
Patente
STECH, Marlitt; HANACK, Katja; MESSERSCHMIDT, Katrin; KUBICK, Stefan: METHOD FOR PRODUCING POLYCLONAL ANTIBODIES USING AN ANTIGENIC COMPOSITION COMPRISING PROTEIN-CONTAINING MEMBRANE VESICLES. PCT/EP2014/002592
STECH, MARLITT; QUAST, ROBERT; WUESTENHAGEN, DOREEN; KUBICK, STEFAN: Verfahren und vorrichtung zur zellfreien proteinsynthese in gegenwart eines caspase-inhibitors. PCT/EP2014/002520
 Fraunhofer-Institut für Zelltherapie und Immunologie, Institutsteil Bioanalytik und Bioprozesse IZI-BB
Fraunhofer-Institut für Zelltherapie und Immunologie, Institutsteil Bioanalytik und Bioprozesse IZI-BB